2019-07-22
几十年来,DNA一直被认为是决定生命遗传信息的核心物质,但是近些年不断的研究表明,生命遗传信息从来就不是基因所能完全决定的,比如科学家们发现,可以在不影响DNA序列的情况下改变基因组的修饰,这种改变不仅影响个体的发育,而且还可遗传给后代。如肿瘤等多种疾病并非仅由基因突变而引起,且与DNA和组蛋白修饰等改变而引起的基因异常表达密切相关,这就是我们今天要分享的一个新领域——表观遗传学。随着基因编辑技术的快速发展,目前不仅可以在DNA水平高效编辑校对,同时也可在不改变基因序列的情况下对基因进行操纵,进一步达到相应基因转录调控目的,成为了精准医疗的研究热点。
何为表观遗传学?
遗传学是指基于基因序列改变所致基因表达水平变化,如基因突变、基因丢失等;而表观遗传学则是指基于非基因序列改变所致基因表达水平变化,如DNA甲基化和染色质构象变化等。2018年发表在国际知名期刊NEJM(The new England journal of medicine)重磅综述中提到,表观遗传学主要包括四大类[1]:
▼ DNA甲基化:它是5'端核苷酸胞嘧啶的共价修饰,通常与基因沉默有关,是表观遗传信息的最清晰的例子;
▼ 核心组蛋白翻译后修饰:如乙酰化,甲基化,磷酸化,泛素化等。每种修饰都与基因活性、基因沉默或活性与非活性基因区域之间的绝缘有关;
▼ 高阶染色质结构:包括通过染色体构象捕获方法揭示的环组织(即用于分析细胞中染色质的高阶组织的技术);有组织的染色质赖氨酸(K)修饰(LOCKs),和涉及多基因区域的核区域的核层结构域(LAD)。
▼ 非编码RNA:如常见的siRNA,piRNA,miRNA及长非编码RNA都属于表观遗传的范畴。
大量研究表明疾病的发生与表观遗传学密切相关,癌症是常见表观遗传疾病的范例。例如甲基化变异性的增加,与白血病和淋巴瘤中侵袭性更强有关,在造血系统肿瘤的结局预测中也有类似的观察结果,慢性淋巴细胞白血病显示出更高的内部甲基化变异性。这种表观遗传变化,特别是表观遗传标记的变异性,可能是有价值的、诊断和预后癌症的工具。如下图为正常细胞和肿瘤细胞在表观遗传上的对比:
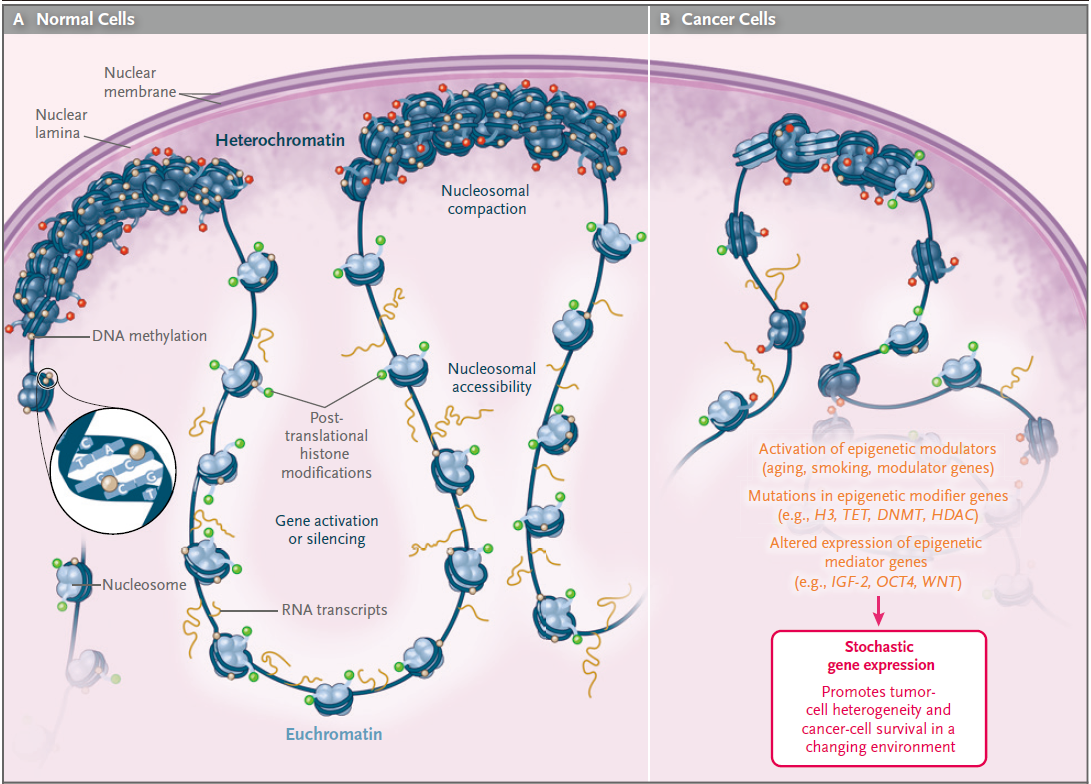
图1. 表观遗传在正常细胞和癌细胞的变化(来源NEJM)
基因编辑如何在表观基因组操控?
基因编辑工具的迅速发展使得在天然染色质环境中有针对性地进行表观基因组编辑和转录调控成为可能,特别是CRISPR/Cas9技术的发展。
优势:能够调控基因的表达而不会造成永久性的DNA损伤,因而不会产生有害突变和脱靶效应;并且表观遗传疗法可以通过同时调节多个基因活性来提供更好的治疗效果,弥补基因治疗存在的不足。
治疗原理:目前,表观遗传修饰和转录调控的设计主要基于核酸酶的结合运用,其中以工程化缺陷型核酸酶(dCas9)的结合最为有效[2]。dCas9是Cas9蛋白的突变体,即Cas9蛋白的RuvC1和HNA两个核酸酶活性区域同时发生突变。因此,dCas9蛋白的内切酶活性完全消失,只保留由gRNA引导进入基因组的能力。
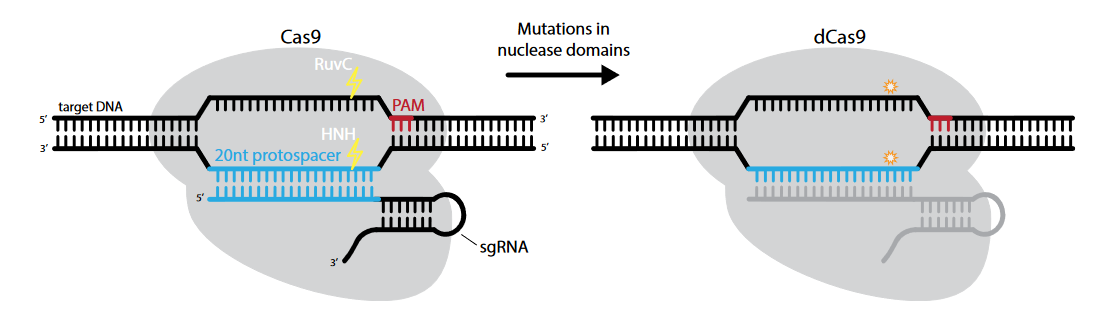
图2. dCas9示意图[2]
CRIPSR-dCas9系统提供了一个研究转录调控和进行表观基因组编辑的平台,其设计原理在于把各种表观调控效应器融合到dCas9上,通过核酸酶的靶向并结合目标DNA的特性实现特定基因组位点上表观基因组的编辑。这些反式调控结构域和蛋白是通过向启动子区域的dCas9靶向位点阻碍RNA聚合酶的结合或募集内源性转录复合物来发挥作用的。以下就是它的两类主要作用模式:
第一类:dCas9融合转录因子或转录原件,通过招募转录因子或修饰因子起到调控作用。以下便是常用的一些基础原件:
▼ 常用的抑制因子:dCas9- KRAB [3-4] 。在这个系统中,dCas9与Kox1的转录阻遏物结构域KRAB(Kruppel-associated box)融合,这种增强的CRISPRi系统依靠KRAB招募各种各样的组蛋白修饰因子,通过形成异染色质的方式可逆地抑制基因表达。
▼ 常用的激活因子:dCas9-VP64[5-6]。VP64是一种RNA结合蛋白,可以与转录因子的转录活化结构域结合,从而提高基因的表达水平。
▼ 除了这些因子之外,dCas9-VPR[7]和dCas9-TV[8]是比dCas9-VP64更有效的转录激活剂。
第二类:dCas9与表观遗传修饰酶来进行融合,直接催化DNA或者组蛋白的表观修饰,调控基因的表达。
dCas9-DNMT3A已被证实可使靶向启动子上的DNA瞬时甲基化[9],从而抑制基因的表达。相比之下,使用dCas9-TET1则可实现启动子的快速和瞬时DNA去甲基化,诱导靶基因的表达上调[9-10]。此外,通过dCas9-PRDM9和dCas9 -DOT1L可以实现组蛋白稳定的甲基化,以重新启动表观遗传相关沉默基因的表达[11]。目前也有研究报道通过dCas9-LSD1诱导增强子的组蛋白脱甲基化,可下调靶基因的表达[12]。通过使用dCas9-P300[13]和dCas9-HDAC3[14],在靶向增强子和启动子区域可实现组蛋白瞬时高效的乙酰化和脱乙酰化,调控基因的表达。各种调控效应器同dCas9的结合极大地扩展了CRISPR在转录调控和表观基因组编辑的应用范围,而相关研究也表明核酸酶的表观基因组编辑具有极高的安全性和特异性,并且没有明显的脱靶效应[3],[15],为体内表观基因修饰和治疗奠定了基础。
细心的读者可能注意到了,CRISPR介导的表观遗传调控与我们之前介绍过的单碱基编辑工具也是有一定的相同之处的。单碱基编辑系统和表观遗传调控系统都是利用了缺陷型的Cas9蛋白(前者是只产生nick而不产生DSB的nCas9,后者是完全失去内切酶活性的dCas9),而且两者都是通过融合蛋白来起到工作效果(前者是融合碱基编辑蛋白,后者是融合激活因子或抑制因子)。下图是一个单碱基编辑与表观基因组编辑的示意图,读者们可以感受一下其中的奥妙。
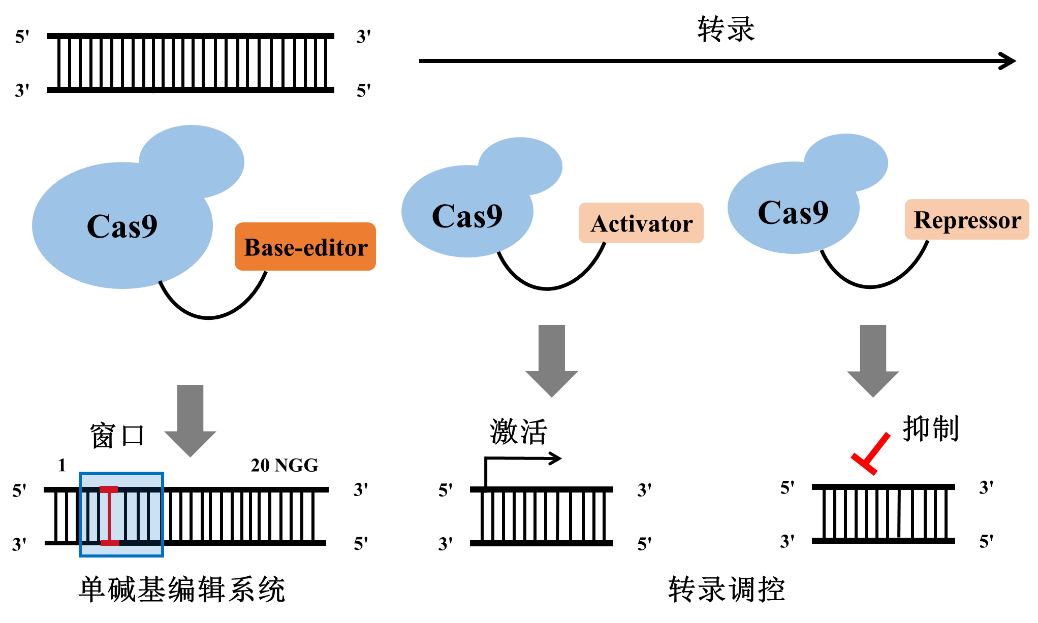
图3. 单碱基编辑与表观基因组编辑示意
在基因治疗上的研究进展
其实,基因组表观遗传修饰的异常与许多疾病的发生与发展息息相关,如生活中常见的代谢紊乱、心血管疾病和癌症等等。基于蛋白编码的基因研究是不足以解释这一类疾病的发生机理的,这些疾病的发病机制主要是基因和环境共同作用的结果。转录调控和表观遗传修饰改变的可逆性为治疗这类疾病提供了新的希望。基因定点修饰技术的发展更为其精准治疗增加了可能。
目前利用转录调控和表观遗传定点修饰技术在体内治疗表观遗传异常引起疾病的研究还屈指可数。接下来小编就带大家回顾一下现有的基因编辑结合表观遗传治疗的代表工作。
1. 常见体细胞遗传疾病
▼ 致病机理:体细胞中由于遗传缺陷导致的疾病。
▼ 代表工作:2017年,一项研究采用了一种gRNA融合MS2-P65-HSF1(MPH)转录激活复合物的策略,将截短的gRNA(dgRNA)和MPH包装到两个单独的AAV载体中,共同递送到表达dCas9的转基因小鼠中。通过靶向激活相应的功能性基因成功地修复了I型糖尿病、急性肾损伤和肌营养不良的疾病表型[16]。
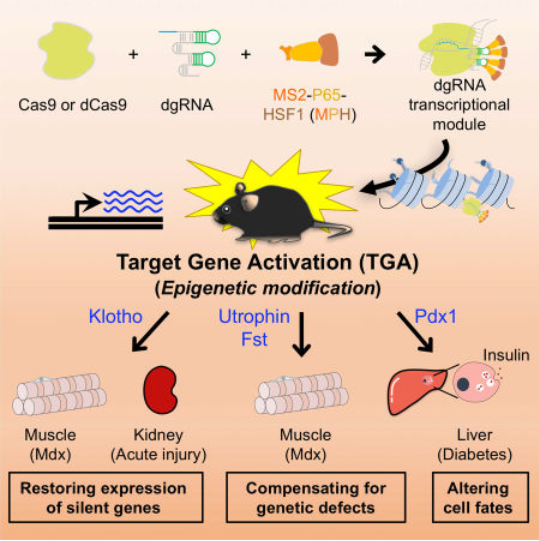
图4. 通过CRISPR介导的表观遗传调控激活体内靶基因进行遗传疾病治疗[16]
2. 神经系统疾病
▼ 致病机理:神经细胞中由于遗传缺陷导致的疾病
▼ 代表工作:同时另一项突破性的工作则使用一种SunTag(dCas9-10xGCN4)系统融合多个拷贝的转录激活蛋白(p65-HSF1),构建了一种Cre依赖性的SunTag-p65-HSF1(SPH)转基因小鼠模型。使用AAV8将Cre和sgRNAs递送到SPH转基因小鼠中,通过激活内源性神经源性转录因子的表达,在小鼠体内成功地实现了星形胶质细胞直接转化为功能性的神经元[17]。这一研究表明,在不需要使用外源重编程因子或转录因子的前提下,通过转录调控的CRISPR系统可以实现特定细胞的重编程或不同细胞类型之间的转分化,为体外细胞基因治疗遗传疾病提供了新的策略。
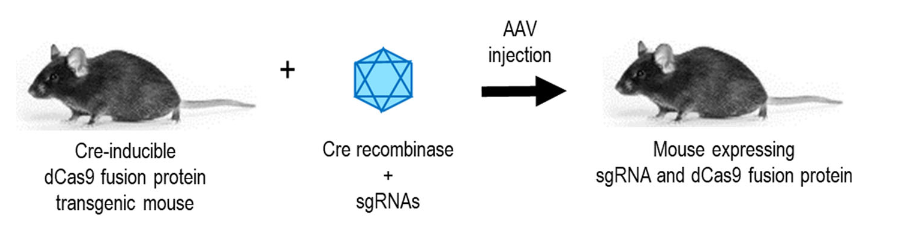
图5. Cre诱导的dCas9小鼠[21]
3. 单倍体剂量不足引起的疾病
▼ 致病机理:单倍剂量不足(haploinsufficiency )指一个等位基因突变后,另一个等位基因能正常表达,但这只有正常水平50%的蛋白质不足以维持细胞正常的生理功能。
▼ 代表工作:近日,Science在线发表了一篇使用CRISPRa系统在小鼠中成功修复一种因单倍剂量不足引起的肥胖。研究人员通过AAV在SIM1基因或MC4R基因部分功能丧失的小鼠脑部递送dCas9-vp64和sgRNA的方式成功激活了SIM1或MC4R蛋白的表达,成功抑制了肥胖的表型[18]。这一策略给罹患单倍体剂量不足引起的疾病患者带来转机。
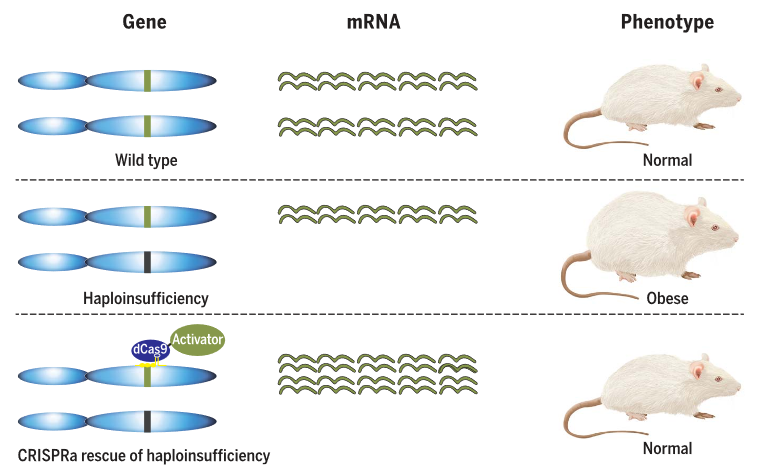
图6. 通过CRISPRa治疗单倍体剂量不足相关疾病[18]
4. 异常甲基化引起的疾病
▼ 致病机理:基因中CpG岛中的5' C经常突变引起高甲基化、羟甲基化等修饰,研究表明这些异常修饰会影响基因的表达调控,最终引起疾病[19]。
▼ 代表工作:脆性X综合征(Fragile X syndrome, FXS)就是一种由FMR1基因5' UTR 区中CGG三核甘酸重复序列扩增突变并高甲基化,使FMR基因沉默而导致的疾病。最近的一项研究通过利用dCas9融合Tet甲基胞嘧啶双加氧酶1(Tet methylcytosine dioxygenase 1, Tet1)转染FXS iPSCs 细胞系,成功靶向诱导FMR基因5' UTR CpG岛去甲基化,为这些因异常甲基化引起的疾病的治疗奠定了基础[20]。
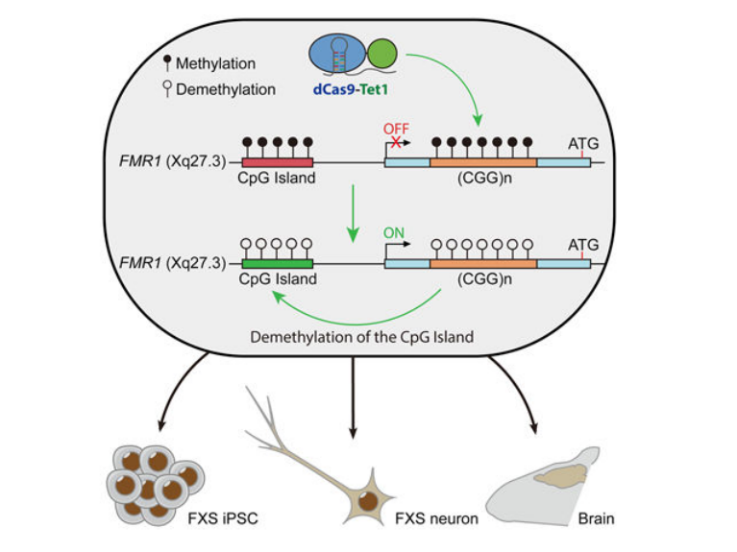
图7. 脆性X综合征相关疾病的表观遗传治疗[20]
怎么样,这次的公众号是不是让大家大开眼界呢?相信通过这四期的公众号,大家已经对基因编辑在疾病治疗中的研究应用有了较为系统的了解,不过“路漫漫其修远兮”,从实验室走到临床还有较为漫长的距离要走。但是大量的实验已经给我们看到了未来疾病治疗的新曙光,相信终有一天,基因编辑治疗的大时代终会到来。
参考文献:
[1]Feinberg AP. The Key Role of Epigenetics in Human Disease Prevention and Mitigation. N Engl J Med. 2018, 378(14): 1323-1334.
[2]Brocken DJW, Tark-Dame M, Dame RT. dCas9: A Versatile Tool for Epigenome Editing. Curr Issues Mol Biol. 2018, 26:15-32.
[3]Thakore PI, D'Ippolito AM, Song L, Safi A, Shivakumar NK, Kabadi AM, Reddy TE, Crawford GE, Gers-bach CA. Highly specific epigenome editing by CRISPRCas9 repressors for silencing of distal regulatory ele-ments. Nat Methods, 2015, 12: 1143-1149.
[4]Gilbert LA, Larson MH, Morsut L, Liu Z, Brar GA, Torres SE, Stern-Ginossar N, Brandman O, Whitehead EH, Doudna JA, Lim WA, Weissman JS, Qi LS. CRISPR-mediated modular RNA-guided regulation of tran-scription in eukaryotes. Cell, 2013, 154(2): 442-451.
[5]Maeder ML, Linder SJ, Cascio VM, Fu Y, Ho QH, Joung JK. CRISPR RNA-guided activation of endogenous human genes. Nat Methods, 2013, 10(10): 977-979.
[6]Perez-Pinera P, Kocak DD, Vockley CM, Adler AF, Kabadi AM, Polstein LR, Thakore PI, Glass KA, Oust-erout DG, Leong KW, Guilak F, Crawford GE, Reddy TE, Gersbach CA. RNA-guided gene activation by CRISPR-Cas9- based transcription factors. Nat Methods, 2013, 10(10): 973-976.
[7]Chavez A, Scheiman J, Vora S, Pruitt BW, Tuttle M, P R Iyer E, Lin S, Kiani S, Guzman CD, Wiegand DJ, Ter-Ovanesyan D, Braff JL, Davidsohn N, Housden BE, Perrimon N, Weiss R, Aach J, Collins JJ, Church GM. Highly efficient Cas9-mediated transcriptional programming. Nat Methods, 2015, 12(4): 326-328.
[8]Li Z, Zhang D, Xiong X, Yan B, Xie W, Sheen J, Li JF. A potent Cas9-derived gene activator for plant and mammalian cells. Nat Plants, 2017, 3(12): 930-936.
[9]Liu XS, Wu H, Ji X, Stelzer Y, Wu X, Czauderna S, Shu J, Dadon D, Young RA, Jaenisch R. Editing DNA methylation in the mammalian genome. Cell, 2016, 167(1): 233-247.
[10]Liu P, Chen M, Liu Y, Qi LS, Ding S. CRISPR-based chromatin remodeling of the endogenous Oct4 or Sox2 locus enables reprogramming to pluripotency. Cell Stem Cell, 2018, 22(2): 252-261.
[11]Cano-Rodriguez D, Gjaltema RA, Jilderda LJ, Jellema P, Dokter-Fokkens J, Ruiters MH, Rots MG. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nat Commun, 2016, 7: 12284.
[12]Kearns NA, Pham H, Tabak B, Genga RM, Silverstein NJ, Garber M, Maehr R. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods, 2015, 12(5): 401-403.
[13]Hilton IB, D'Ippolito AM, Vockley CM, Thakore PI, Crawford GE, Reddy TE, Gersbach CA. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol, 2015, 33(5): 510-517.
[14]Kwon DY, Zhao YT, Lamonica JM, Zhou Z. Locus specific histone deacetylation using a synthetic CRISPRCas9-based HDAC. Nat Commun, 2017, 8: 15315.
[15]Mendenhall EM, Williamson KE, Reyon D, Zou JY, Ram O, Joung JK, Bernstein BE. Locus-specific editing of histone modifications at endogenous enhancers. Nat Biotechnol, 2013, 31(12): 1133-1136.
[16]Liao HK, Hatanaka F, Araoka T, Reddy P, Wu MZ, Sui Y, Yamauchi T, Sakurai M, O'Keefe DD, Núñez-Delicado E, Guillen P, Campistol JM, Wu CJ, Lu LF, Esteban CR, Izpisua Belmonte JC. In vivo target gene activation via CRISPR/Cas9-mediated trans-epigenetic modulation. Cell, 2017, 171(7): 1495-1507.
[17]Zhou H, Liu J, Zhou C, Gao N, Rao Z, Li H, Hu X, Li C, Yao X, Shen X, Sun Y, Wei Y, Liu F, Ying W, Zhang J, Tang C, Zhang X, Xu H, Shi L, Cheng L, Huang P, Yang H. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice. Nat Neurosci, 2018, 21(3): 40-446.
[18]Matharu N, Rattanasopha S, Tamura S, Maliskova L, Wang Y, Bernard A, Hardin A, Eckalbar WL, Vaisse C, Ahituv N. CRISPR-mediated activation of a promoter or enhancer rescues obesity caused by haploinsufficiency. Science, 2018.
[19]Xu Y, Wu F, Tan L, Kong L, Xiong L, Deng J, Barbera AJ, Zheng L, Zhang H, Huang S, Min J, Nicholson T, Chen T, Xu G, Shi Y, Zhang K, Shi YG. Genome-wide regulation of 5hmC, 5mC, and gene expression by Tet1 hydroxylase in mouse embryonic stem cells. Mol Cell, 2011, 42(4): 451-464.
[20]Hayashi ML, Rao BS, Seo JS, Choi HS, Dolan BM, Choi SY, Chattarji S, Tonegawa S. Rescue of Fragile X Syndrome Neurons by DNA Methylation Editing of the FMR1 Gene. Cell, 2018, 172(5): 979-992.e6.
[21]Lau CH, Suh Y. In vivo epigenome editing and transcriptional modulation using CRISPR technology. Transgenic Res. 2018, 27(6):489-509.